61 KiB
🍄 Projet de groupe 2024 sur la reconnaissance de champignons 🍄
Auteurs : Heuzef, Yvan Rolland, Viktoriia Saveleva, Florent Constant
Rendu N°1 : Rapport d’EDA
Date : 06/2024
1. Introduction
La compréhension des champignons est cruciale pour la préservation de la biodiversité, la santé humaine et l’agriculture durable.
Les champignons ne sont pas des plantes, bien que leur apparente immobilité puisse le faire penser. Une distinction simple est que les champignons ne font pas de photosynthèse, contrairement à une majorité de plantes. En fait, dans l’arbre de la vie, les champignons sont plus proches des animaux que des plantes bien que leur mode de nutrition, paroi cellulaire, reproduction, les distingues également nettement des animaux.
L’arbre de la vie, qui représente la parenté entre les organismes vivants, peut être découpé en six règnes. Les champignons représentent rien qu'à eux le règne fongique, qui rassemblerait hypothétiquement jusqu'à 5 millions d’espèces de champignons. Parmi toutes ces espèces, environ seulement 120 000 ont été nommées et “acceptées” par la communauté scientifique en 2017.
La reconnaissance de champignons représente un défi dans le domaine de la vision par ordinateur. En effet, les critères biologiques et le peu d’espèce référencés limite à une reconnaissance peu fiable et sur un échantillon insignifiant si l’on souhaite étudier l’ensemble du règne fongique.
La classification classique des vivants est schématisée ainsi :
| Classification classique des règnes |
|---|
| Animal |
| Plante |
| Champignon (Fungi) |
| Protiste |
| Bactérie |
| Archée |
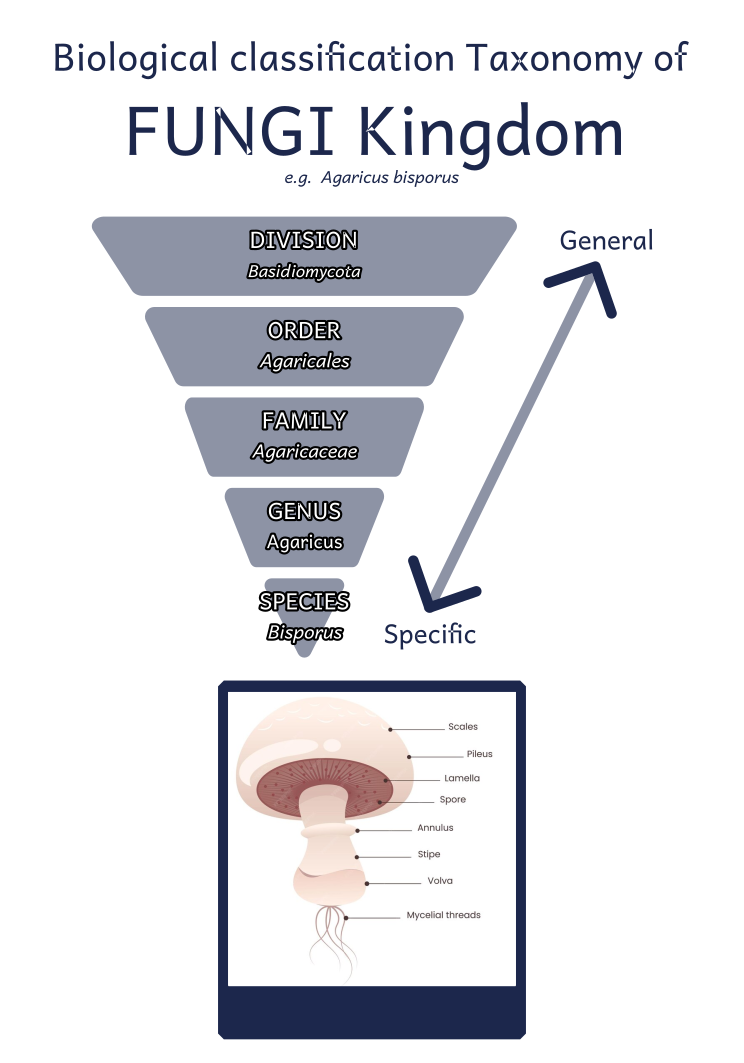
Et les principaux rangs taxonomiques ainsi (ici, un exemple de classification du très connu “Champignon de Paris") :
On y voit que les champignons sont classés (du plus général au plus spécifique) en divisions, ordres, familles, genres et espèces.
L'état de l’art nous apprend que la reconnaissance des champignons au sens large ne sera possible que sur un échantillon très faible du règne fongique, mais ce n’est pas tout, en effet, la vision par ordinateur effectue un balayage des images matricielle pour comparer les similitudes pour chaque pixel avec des images déjà labellisé, ainsi, nous dépendons de la qualité des sources de donnés, qui ne représentent qu’un échantillon des ~120 000 espèces scientifiquement nommées sur un total pouvant aller jusqu'à 5 millions d’espèces théorique.
Il existe également la distinction entre macro-champignons et micro-champignons, qui se base sur une combinaison de caractéristiques morphologiques, cellulaires, reproductives, écologiques et économiques. L’identification précise des champignons exige des connaissances approfondies en mycologie. Par ailleurs les différentes sources alertent quand à la difficulté de l’identification d’une espèce se basant uniquement sur l’aspect visuel.
À ce jours, il existe approximativement 35000 genres et de champignon sur terre et certain peuvent compter jusqu'à des milliers espèces nommés, tandis que d’autre peuvent n’en compter qu’une seul.
Une analyse visuelle des différents rangs taxonomiques sur des échantillons de photos extraite de Mushroom Observer nous laisse penser que c’est au niveau de l’espèce que nous pouvons observer les plus de traits caractéristiques en vue de réaliser une identification visuelle :
Visuel par ordre :
Des champignons appartenant à l’ordre des PEZIZALES :
Visuel par famille :
Des champignons appartenant à la famille des RUSSULACEAE :
Visuel par genre :
Des champignons du genre CANTHARELLUS :
Visuel par espèce :
Des champignons appartenant à l’espèce HYPHOLOMA LATERITIUM :
C’est également communément le niveau d’identification recherché car c’est au niveau de l’espèce que sont définies les principales propriétés d’un champignon, telles que la comestibilité.
Nous constatons également que les champignons peuvent avoir des formes si variées que deux champignons de la même espèce peuvent avoir un aspect très différent (notamment en fonction de l’age), alors que deux champignons d’espèces différentes peuvent afficher une très forte ressemblance.
Variétés de formes dans une même espèce
Pour illustration, deux champignons de l’espèce Coprinus comatus mais visuellement très différents :
Confusions possibles
De même deux champignons de genres différents visuellement difficiles à distinguer, ici Clytocibe nébuleux et Entolome livide :
2. Objectif
Ce premier niveau de connaissance de la problématique d’identification visuelle d’un champignon nous permet de distinguer trois difficultés majeures du domaine :
-
L’immense quantité d’espèces existantes, la proximité visuelle importante existant entre certaines espèces et la différence morphologique pouvant exister au sein d’une même espèce.
-
La quantité et la qualité des données disponibles seront déterminantes pour obtenir un modèle performant.
-
Selon nos propres capacités et le temps disponible pour la réalisation du projet, nous pourrons fixer différents niveaux d’objectifs à atteindre pour notre projet, l’essentiel restant l’aspect pédagogique et l’acquisition de compétences.
L’objectif primaire est d’entraîner un modèle pour la reconnaissance des champignons. Pour atteindre cet objectif, il faudra suivre les étapes suivantes :
-
Analyser la taxonomie et définir le niveau sur lequel nous concentrer
-
Analyser les données disponibles
-
Trier et filtrer les données
-
Data augmentation (créer de nouveaux échantillons d’entraînement en appliquant diverses transformations aux images existantes)
-
Prétraitement des données
-
Poursuivre avec des techniques de deep learning
Nous pourrons donc travailler à entraîner un modèle capable d’identifier un nombre plus ou moins grand d’espèces avec le plus de précision possible. Le niveau de difficulté pourra donc être modulé selon le nombre d’espèces introduites mais aussi la ressemblance visuelle entre les différentes espèces introduites.
Nous pourrons également envisager différentes approches, par exemple entraîner et utiliser un modèle pour faire du “boxing”, générer des données artificielles par des transformations des images de notre jeu de données, essayer de quantifier le volume d’images nécessaire pour l’obtention d’un certain niveau de performances ...
3. Sources de données identifiées
Les ensembles de données contenant des champignons sont largement utilisés pour l’entraînement des algorithmes de machine learning et de deep learning. Divers ensembles de données sont disponibles en accès libre pour différentes finalités.
UC Irvine Mushroom Database (https://archive.ics.uci.edu/dataset/73/mushroom) comprend 8 124 enregistrements de données et 22 attributs. Chaque espèce de champignon est identifiée comme une classe de champignons comestibles ou toxiques. Ces données sont réparties en 4 208 champignons comestibles et 3 916 champignons toxiques. De nombreuses approches différentes sont présentées dans la littérature pour travailler avec ce type de caractérisation des champignons basée sur les caractéristiques physiques (pas d’images). Cependant, dans ce travail, nous nous concentrons principalement sur la reconnaissance d’images, notre attention se portant sur les ensembles de données d’images.
Mushroom Observer
Mushroom Observer est un site web où les gens peuvent télécharger des observations de champignons. Ces observations incluent différentes informations sur l’espèce observée, comme le nom, l’emplacement, et la certitude concernant l’espèce sur les images soumises. Le site est basé sur des photos prises par un grand nombre d’individus qui ne sont pas nécessairement des experts dans ce domaine. La certitude des étiquettes de classe, donnée par la communauté est sur une échelle continue de 1 à 3 (où 3 représente le plus haut niveau de certitude).
L’analyse des images de Mushroom Observer montre deux problèmes principaux liés à :
- la qualité des images. Il y a beaucoup d’images qui ne sont pas exploitables : schémas, clichés microscopiques, etc ...
Exemples de photos inexploitables
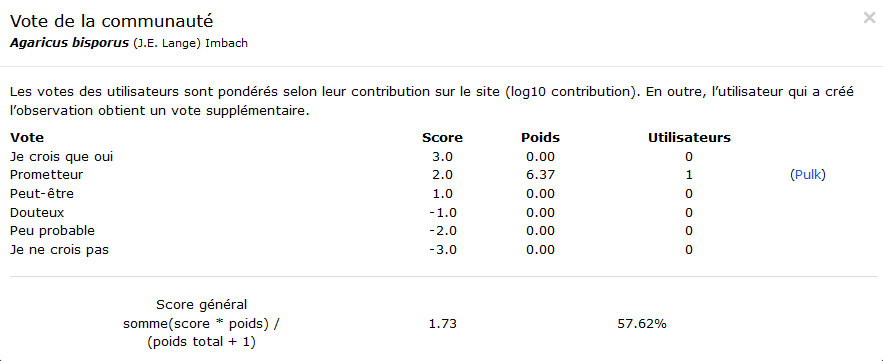
- le niveau de fiabilité de l’attribution de classe. Le système de vote pour la classification des champignons ajoute de l’incertitude dans l’attribution de classe.
Ainsi, la base de données ne peut pas être utilisée telle quelle à partir du site web et doit être filtrée.
MO106 Database
En analysant la littérature utilisant l’ensemble de données Mushroom Observer, nous avons trouvé une base de données MO106 disponible en accès libre où les auteurs ont sélectionné 106 classes de champignons de Mushroom Observer en utilisant les critères suivants : espèces ayant au moins 400 images, images avec certitude ≥ 2. De plus, pour filtrer automatiquement les images afin d’obtenir une image correcte de champignon (sans objets supplémentaires ou sans champignons), les auteurs ont formé un modèle CNN spécifique.
Cela a abouti à un ensemble de données MO106 contenant 29 100 images réparties en 106 classes. La plus grande classe compte 581 éléments, la plus petite 105, avec une moyenne de 275. Les images, disponibles gratuitement pour le téléchargement, ont des tailles variant entre 97 × 130 (plus petite surface) et 640 × 640 (plus grande surface).
Pour une observation nous obtenons :
- Photos
- Genre et espèces
Mushrooms classification - Common genus’s images
Dataset de champignons basés sur des images.
Cet ensemble de données contient 9 dossiers d’images des genres de champignons les plus communs du nord de l’Europe (Agaricus, Amanita, Boletus, Cortinarius, Entoloma, Hygrocybe, Lactarius, Russula et Suillus). Chaque dossier contient entre 300 et 1 500 images sélectionnées de genres de champignons. Les étiquettes correspondent aux noms des dossiers. Des codes de classification utilisant cet ensemble de données sont également disponibles.
L’avantage de cette base de données par rapport à Mushroom Observer est que la classification a été vérifiée par la société de mycologie d’Europe du Nord, qui a fourni les sources des champignons les plus communs de cette région et a vérifié les données et les étiquettes.
Pour une observation nous obtenons :
- Photos
- Genre et espèces
MycoDB
Le site mycodb.fr nous permet d’acquérir des caractéristique précises d’un champignon identifié via un nom binominal, pour une observation nous obtenons :
- Photos
- Division - Classe - Ordre - Famille
- Synonymes
- Chapeau
- Lames
- Stipe
- Saveur
- Odeur
- Couleur de la sporée
- Ecologie
- Comestibilité
- Références bibliographiques
Wikipedia
Wikipédia reste une source d’information très complémentaire et souvent exhaustive pour en apprendre plus sur un genre ou une espèce de champignon.
Conclusion
Après identification de ces différentes sources de données nous concluons que Mushroom Observer sera celle qui sera la plus exploitable pour obtenir des données de qualité. Le site dispose d’une API permettant un accès à la quasi totalité des données, permettant d’obtenir une visualisation précise du nombre d’espèces répertoriées ainsi que du nombre d’observations et d’images associées à chaque espèce.
Par ailleurs le jeu de données MO106 déjà extraites de Mushroom observer pourrait être une source inintéressante car déjà prête à l’emploi bien que la qualité des images sélectionnée échappe à notre contrôle. Cela pourra par exemple donner lieu à un comparatif de précision des résultats en fonction de la qualité des images en entrée.
4. Exploration approfondie des données disponibles sur Mushroom observer
Le principal avantage de Mushroom observer est qu’il met à disposition une API permettant d’accéder a des données structurées issues de sa base. Ces données nous permettrons de faire une analyse qualitative et quantitative des images disponibles. Les données ont été téléchargées au format CSV et sont présentes sur le dépôt du projet.
Principales tables
table names
Cette table contient l’arborescence des nommages disponibles sur le site, répartis en niveaux (rangs) de la manière suivante :
- forme
- variété
- sous-espèce
- espèce
- stirpes
- sous-section
- section
- sous-genre
- genre
- famille
- ordre
- classe
- phylum
- regne
- domaine
- groupe
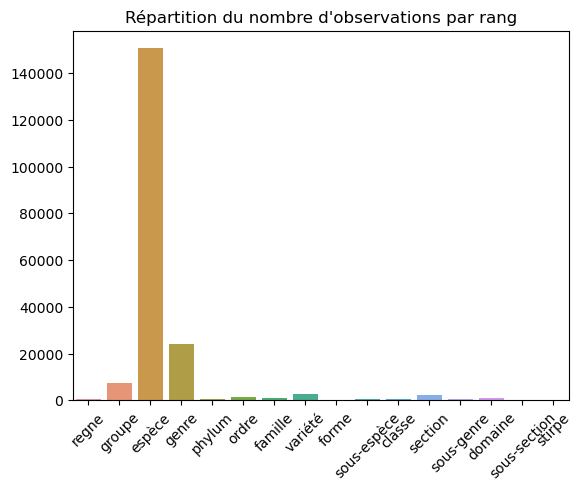
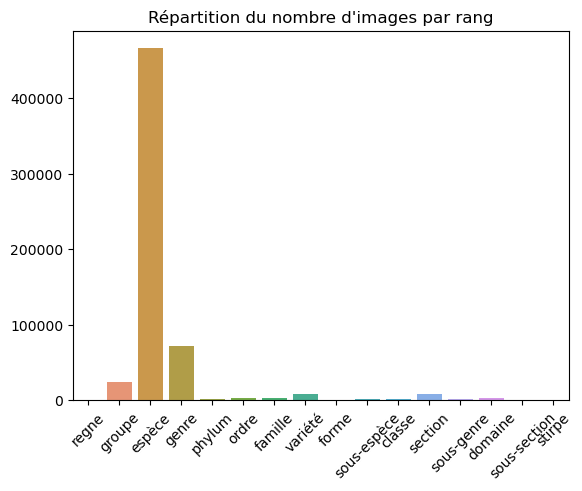
Nous observons par exemple que le site répertorie à ce jour 56161 espèces.
table observations
Cette table permet de quantifier le nombre d’observations réalisées pour chaque espèce mais aussi de qualifier la fiabilité de ces observations : le site offrant un système participatif, l’identification des champignons est soumise au vote des utilisateurs du site. La note de confiance concernant l’identification d’une observation varie de -3 à 3. Après évaluation du nombre d’observation disponible nous choisirons de ne conserver que celles dont le score de confiance est >= 2.
Le graphique montre que le jeu de donnée comprends environs 150k observations rattachées à une espèce avec un niveau de confiance >= 2.
table images_observation
Cette table liste les images rattachées à chaque observation. Sans surprise les quantités d’images rattachées à chaque rang sont proportionnelles a la quantité d’observations. Nous constatons que pour notre sélection de critères environs 500k images sont disponibles.
Agrégation par espèces
Nous savons que nous devons disposer d’une quantité minimale d’images pour chacune des espèces sur lesquelles nous souhaitons entraîner notre modèle. Bien que cette quantité soit encore à définir précisément, nous estimons que 150-250 images serait une base de départ viable. Nous constatons aussi que la moitié environs des images est exploitable, le reste n'étant pas directement des photographie des spécimens de champignons.
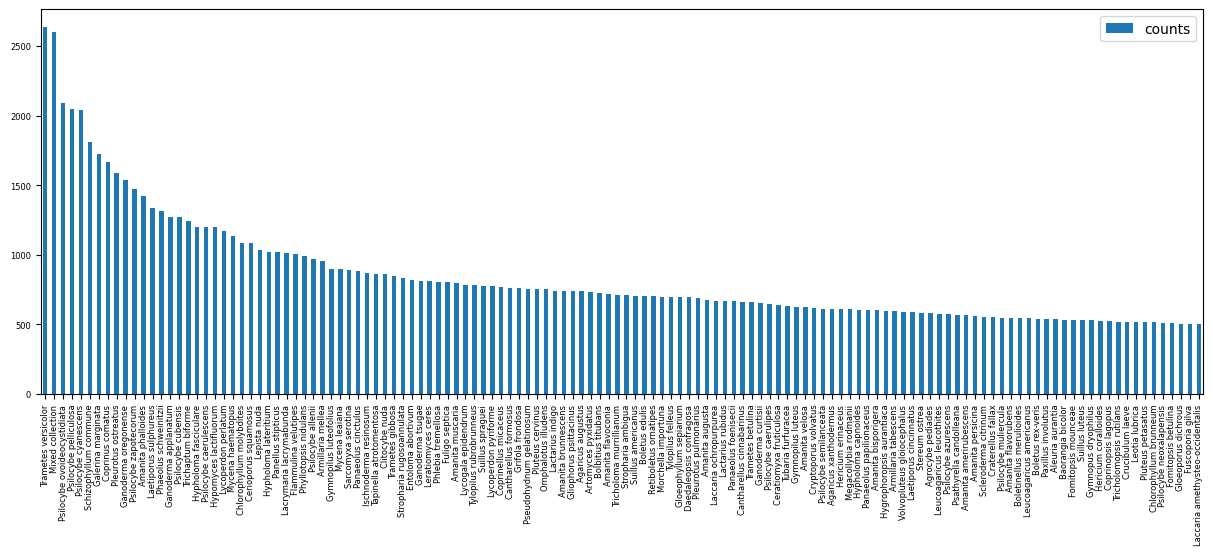
Un second filtrage est effectué pour ne sélectionner que les espèces qui disposent d’au moins 500 photos sur le site. Nous pouvons donc compter disposer de données suffisantes pour 129 espèces.
Sélection finale des images
Nous avons identifié le besoin de filtrer manuellement les images avant pré-traitement pour exclure celle qui ne sont pas exploitables (schémas, clichés microscopiques, etc ...). Nous avons donc réalisé un outil proposant une interface permettant de réaliser le tri de manière relativement efficace. Nous pourrons constituer un jeu de données d’images triée plus ou moins important selon les besoins et le temps disponible au fil de l’avancée du projet.
L’outil est disponible sur le dépôt du projet.
Une fois la sélection effectuée, nous pouvons alors exécuter le script de webscraping nous permettant de télécharger les photos sélectionnés (cf: Annexes). Pour certains champignons, nous avons plus d’une photo. Nous nous concentrons uniquement sur la première (le script sélectionne uniquement la première image de la série).
A la date de rédaction de ce rapport nous avons réuni 2282 images appartenant à 14 espèces différentes.
Organisation des données
Le stockage des données, (dans espace de stockage privé), est structurée ainsi :
data
├── LAYER0
│ ├── MO
│ ├── MO
│ │ └── dataset.csv
│ └── MO_106
│ ├── MO_106
│ ├── class_stats.csv
│ ├── dispersion.csv
│ └── image_stats.csv
├── LAYER1
│ └── MO
│ └── MO
└── LAYER2
└── MO
└── MO
Cette configuration nous permettra ultérieurement de fournir la base d’image MO ou MO_106 à nos différent modèles facilement.
5. Pré-traitement des données
Choix des outils de preprocessing
Pour le prétraitement des données, nous avons sélectionné des outils spécifiques de preprocessing. Ensuite, nous appliquons un second traitement avec le modèle YOLOv5 (You Only Look Once version 5), qui permet une détection rapide et précise des champignons en les identifiant par encadrement (cf: Annexes).
Cela nous permet d’obtenir des images précise indispensables pour les étapes suivantes d’entraînement de modèle. Cet outil n'étant pas parfait, nous compensons les échecs de celui-ci avec un outil d’encadrement manuel développé pour l’occasion (cf: Annexes).
Afin de préparer nos images pour les entraînements à venir, nous appliquons les méthodes conventionnelles et récurrentes pour le CNN.
Redimensionnement des images
La réduction des images à une taille de 224 x 224 pixels est couramment utilisée dans les architectures de réseaux de neurones convolutionnels (CNN) pour plusieurs raisons pratiques et techniques tel que la standardisation, la gestion de la mémoire et des ressources computationnelles, la comparaison avec les modèles pré-entraînés et la capture des caractéristiques importantes.
Enrichir le jeu de données
Nous réalisons une étape de ré-échantillonnage afin d’augmenter le volume de données d’entraînement, pour les futurs modèles que nous testerons. Cela nous permettra d’améliorer la précision des modèles.
Nous avons réalisé un script exploitant ImageDataGenerator de la librairie tensorflow.keras.preprocessing.image (cf: Annexes).
Nous effectuons ainsi l’augmentation des données avec les techniques suivantes :
-
Rotations
-
Retournement horizontal
-
Retournement vertical
-
Cisaillements
Cela permet de créer de nouveaux échantillons d’entraînement à partir des images existantes, augmentant ainsi la robustesse et la capacité de généralisation de notre modèle.
 Exemple d’images enrichies générée depuis une unique image source, à l’aide de la classe ImageDataGenerator de Keras.
Exemple d’images enrichies générée depuis une unique image source, à l’aide de la classe ImageDataGenerator de Keras.
6. Conclusion
Ce rapport nous permet d’avoir un aperçu de la complexité de la reconnaissance de champignons, mettant en lumière les défis et les opportunités qui se présentent dans ce domaine. À travers une exploration détaillée de la taxonomie, des sources de données disponibles et des étapes de prétraitement des données, nous avons identifié les différentes options à explorer pour atteindre notre objectif de développement d’un modèle de reconnaissance de champignons fiable.
L’analyse a révélé plusieurs défis majeurs, notamment la grande diversité des espèces de champignons, la variabilité morphologique au sein d’une même espèce, et la qualité variable des données disponibles, nécessitant ainsi des stratégies de filtration et de prétraitement rigoureuses. Cependant, nous avons également identifié des sources de données prometteuses, qui offrent des ensembles de données volumineux pour l’entraînement de modèles de reconnaissance.
Enfin, nous avons établi un plan d’action clair, comprenant l’analyse approfondie des données disponibles, le prétraitement des images, et l’enrichissement du jeu de données par des techniques d’augmentation. Ces étapes préliminaires posent les fondations nécessaires pour le développement ultérieur de modèles de deep learning, qui seront essentiels pour la reconnaissance précise des champignons.
7. Annexe
- Scripts de webscraping, d’analyse du site Mushroom Observer et de sélection des donnés
- Script de sélection automatique avec YOLOv5
- Outil d’encadrement manuel MPBS
- Script d’oversampling
🍄 Projet de groupe 2024 sur la reconnaissance de champignons 🍄
Auteurs : Heuzef, Yvan Rolland, Viktoriia Saveleva, Florent Constant
Rendu N°2 : Modelisation
Date : 10/2024
1. Introduction
Dans le cadre du projet de reconnaissance de champignons, nous abordons un problème de deep learning qui s’apparente principalement à une tâche de classification.
L’objectif est de classifier différentes espèces de champignons en fonction de leurs caractéristiques visuelles, ce qui s’inscrit dans le domaine de la reconnaissance d’image.
Pour évaluer la performance des modèles développés, nous utilisons principalement la métrique de l’accuracy (précision), car elle permet de mesurer le pourcentage de classifications correctes effectuées. Cette métrique est particulièrement adaptée pour ce projet, car elle fournit une évaluation claire et directe de l’efficacité du modèle à distinguer les différentes espèces de champignons.
Dans ce rapport, nous décrivons nos démarches, réflexions et erreurs. Nous analysons également l’effet de la détéction et de l’augmentation des données sur les résultats de nos entrainements.
Un premier model naïf LeNet est utilisé pour l’expérimentation, puis finalement des algorithmes de transfert learning sont adoptés pour leur efficacités.
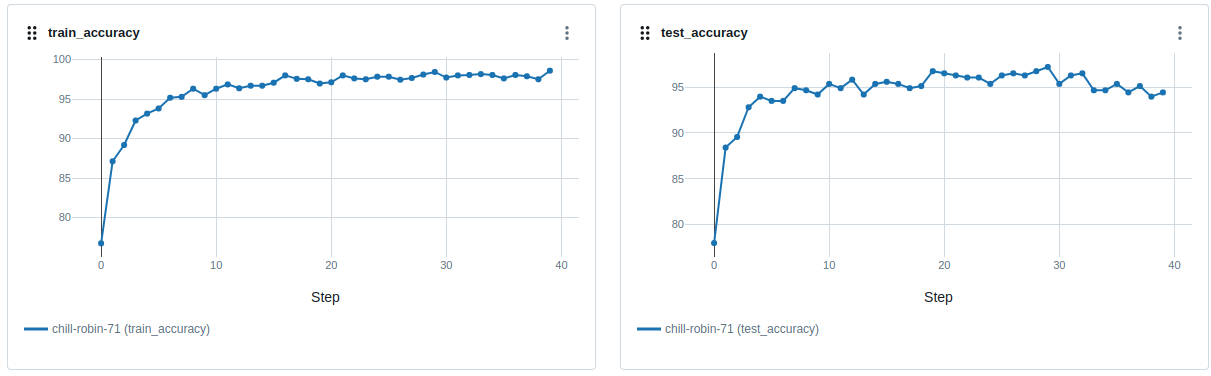
La comparaison des résultats est effectuée en introduisant la partie MLflow dans les algorithmes, ce qui nous permet de suivre la traçabilité et de faciliter l'échange et la comparaison des résultats.
2. Pré-traitement des données
Première approche
Pour rappel, le stockage des données se fait comme suit :
data
├── LAYER0
│ ├── MO
│ ├── MO
│ │ └── dataset.csv
│ └── MO_106
│ ├── MO_106
│ ├── class_stats.csv
│ ├── dispersion.csv
│ └── image_stats.csv
├── LAYER1
│ └── MO
│ ├── MO
│ ├── dataset.csv
└── LAYER2
└── MO
├── MO
├── dataset.csv
└── names.csv
La répartion de ces données intervient dans le but précis de s’assurer de la qualité des donnés avant l’apprentissage pour optimiser les résultats.
LAYER0 : Obtenu par une sélection manuelle et un webscraping, ce qui nous a permis de constituer un dataset comportant 23 classes. L’objectif était d’avoir au moins une centaine de photos par classe. Dans le dossier MO, les photos extraites du site Mushroom Observer. Dans le dossier MO_106, les photos extraites par Webscraping du site : https://www.mycodb.fr/ (utilisées uniquement pour des tests).
LAYER1 : Lancement de la detection effectuée par YoloV5 (boxing), nous perdons environ 60% des images qui n’ont malheureusement pas été detectée par YoloV5. L’objectif est d’obtenir une base de donnée contenant des images de champignons les plus précises. La base de donnée étant l'élément le plus important pour l’apprentissage, il nous apparaissant pertinent de procéder par une détection et un boxing, focus sur le champignon pour limiter le bruit.
Le traitement effectue également des modifications sur l’image nécessaire au Deep Learning : redimensionnement en 224 X 224 px selon les coordonnées du rectangle de détection.
LAYER2 : Créé suite à une augmentation des données.
Cela entraînerait une précision excellente (>0.99) dès la première époque sans optimisation, ce que nous souhaitons éviter.
La séparation initiale garantie que les données d’entraînement et de validation sont distinctes, permettant une évaluation plus précise et une généralisation correcte du modèle.
En résumé, LAYER2 représente les données d’entraînement augmentées, tandis que les ensembles de validation et de test restent intacts et non modifiés pour une évaluation juste et précise du modèle. La figure ci-dessous montre schématiquement la structure des couches.
Deuxième approche
Nos premiers essais montre des performances absolument incroyables, cependant, ceci s’explique par une erreur dans notre approche.
En effet, si nous procédons d’abord par l’augmentation des données puis la division, les ensembles de validation contiendrons des images trop proches de l’entrainement, car simplement modifiées par l’augmentation des données.
À ce stade, il est nécessaire d’effectuer l’inverse, en effectuant l’augmentation des donnés exclusivement sur le jeu d’entrainement divisé en amont, sans toucher au jeu de validation et de test.
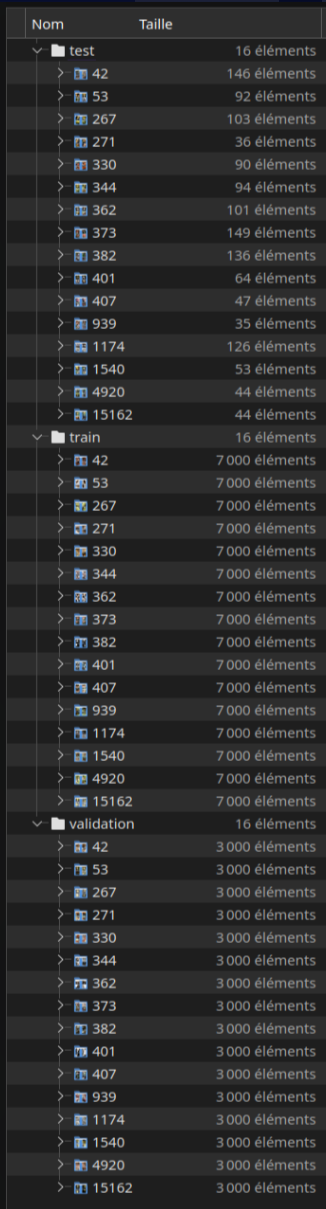
Notre nouvelle arboresence se présente donc ainsi :
data
├── LAYER0
│ ├── dataset.csv
│ └── MO
│ ├── 1174
│ ├── 15162
│ ├── 1540
│ ├── (...)
│ └── 939
├── LAYER1
│ └── MO
│ ├── 1174
│ ├── 15162
│ ├── 1540
│ ├── (...)
│ └── 939
└── LAYER2
├── MO
│ ├── test
│ │ ├── 1174
│ │ ├── 15162
│ │ ├── 1540
│ │ ├── (...)
│ | └── 939
│ ├── train
│ │ ├── 1174
│ │ ├── 15162
│ │ ├── 1540
│ │ ├── (...)
│ | └── 939
│ └── validation
│ ├── 1174
│ ├── 15162
│ ├── 1540
│ ├── (...)
│ └── 939
└── names.csv
Nous prenons à ce stade la décision de ne plus effectuer la detection des champignons via le model YoloV5 pour 2 raisons :
-
La quantitée d’image brutes perdues de 60% est trop importante.
-
Nous ne constatons pas d’amélioration des performances suite à cette detection.
Les données de notre base de donnés “MO” seront divisées un jeu d’entrainement, de validation et de test (directement opéré par le code modèle).
Finalement, nos modèles entrainés seront évalués sur le jeu de test afin de les optimisers pour obtenir la meilleur précision possible.
3. Algorithmes de Deep Learning sélectionnés et Optimisation
Les champignons présentent une diversité visuelle significative, avec des variations subtiles de forme, de couleur, et de texture. Les algorithmes de Deep Learning, notamment les réseaux de neurones convolutifs (CNN), sont particulièrement efficaces pour extraire et apprendre des caractéristiques pertinentes à partir d’images, ce qui en fait l’approche idéale pour identifier correctement les différentes espèces de champignons.
Une première expérimentation est effectuée avec un modèle naïf LeNet, ce dernier permet d’obtenir des résultats interessants, bien que moindre face aux méthodes de Transfert learning, qui offres des performances nettement supérieurs.
En effet, c’est une stratégie clé dans notre approche. En utilisant des modèles pré-entraînés sur de vastes ensembles de données comme ImageNet, nous avons pu adapter ces modèles à notre problème spécifique de reconnaissance des champignons, ce qui a considérablement amélioré les performances des modèles.
Pour cette tâche, nous avons testé plusieurs architectures : VGG16, EfficientNetB1, ResNet50.
Finalement, nous décidons de concevoir un modèle avec une architecture sur mesure: JarviSpore. Ce dernier est partagé sur HuggingFace: https://huggingface.co/YvanRLD
3.1. VGG16
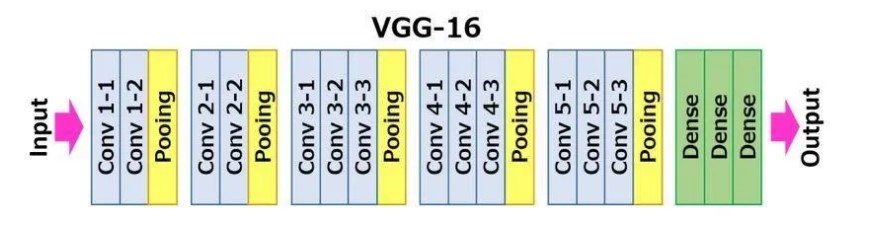
VGG16 est une architecture de réseau de neurones convolutifs (CNN) développée par l'équipe Visual Geometry Group de l’Université d’Oxford. Ce modèle se distingue par sa structure simple et uniforme, composée de 16 couches profondes dont 13 couches convolutives et 3 couches entièrement connectées. Grâce à sa conception, VGG16 a démontré une grande efficacité dans la classification d’images, comme en témoigne ses performances remarquables lors de la compétition ImageNet 2014. Dans le cadre du transfer learning, VGG16 est fréquemment utilisé avec des poids préentraînés sur des ensembles de données étendus tels qu’ImageNet. Cette stratégie permet d’adapter les caractéristiques apprises par le modèle à des tâches spécifiques avec des ensembles de données plus petits, offrant ainsi une précision accrue même avec des données limitées.
Le “16” dans VGG16 fait référence aux 16 couches qui ont des poids. Dans VGG16, il y a treize couches convolutives, cinq couches de max pooling, et trois couches denses, ce qui fait un total de 21 couches, mais seulement seize couches avec des poids, c’est-à-dire des couches avec des paramètres apprenables.
VGG16 prend en entrée un tenseur de taille 224 x 224 px avec 3 canaux RGB. La particularité de VGG16 est qu’au lieu d’avoir un grand nombre d’hyperparamètres, le modèle se concentre sur des couches de convolution avec des filtres de 3x3 avec un stride de 1, en utilisant systématiquement un padding constant et des couches de max pooling avec des filtres de 2x2 et un stride de 2.
Les couches de convolution et de max pooling sont disposées de manière cohérente tout au long de l’architecture. La couche Conv-1 possède 64 filtres, Conv-2 en a 128, Conv-3 en a 256, et Conv-4 et Conv-5 ont 512 filtres chacun.
Trois couches entièrement connectées (FC) suivent une pile de couches convolutives : les deux premières ont chacune 4096 canaux, et la troisième effectue une classification ILSVRC à 1000 classes, contenant ainsi 1000 canaux (un pour chaque classe). La couche finale est la couche softmax.
Les entrainement réalisés
De nombreux entrainements en transfert learning ont été réalisés en utilisant les poids imagenet disponibles directement via keras. Ces entrainements ont permis d’expérimenter deux architectures distinctes ainsi que l’influence de certains hyperparametres :
La première série d’essai a été réalisée avec une architecture comportant une seule couche dense pour la classification. Les expérimentations menées consistaient à faire varier le batch size et le taux d’augmentation des données. Les résultats semblent indiquer globalement que le batch size influence de manière importante la precision du modèle. Dans les essais menés plus la taille du batch size était importante, plus la précision était grande. Les limite de mémoire de la machine de test n’ont pas permis d’expérimenter un batch size au delà de 128. Par ailleur on note que l’augmentation de données n’a quasiement aucune influence sur le résultats. Cette architecture a permis d’obtenir des scores compris entre 66% et 76% de précision.
La seconde serie d’essais a été réalisée sur une architecture comportant trois couches denses, comme le modèle vgg16 original mais entrecoupées de couches dropout. Les expérimentations menées, en plus de faire varier le batch size et le taux d’augmentation des données, consistaient à faire varier le nombre de cannaux et et le taux de dropout des différentes couches.
Les combinaisons suivantes ont été testées :
-
Batch size 32 et 64
-
Nombre de cannaux dans les 2 premières couches de classification: 64,128,256,512,1024,2048
-
Taux de dropout entre chaque couches: 0%, 10%, 20%
Cette architecture a permis d’obtenir des scores compris entre 70% et 77% de précision.
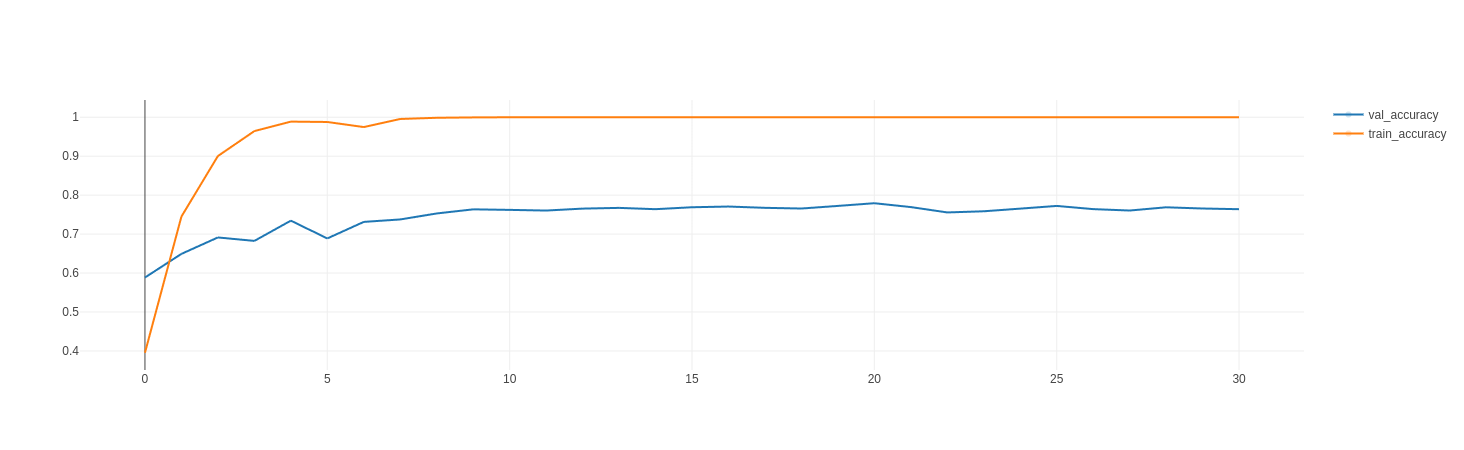
Ces entrainements ont révélé une très forte tendance du modèle à faire du sur-apprentissage sur notre jeu de données, avec une précision sur les données de test rapidement à 100% quelque soit l’architecture et les paramètres employés:
3.2. EfficientNetB1
3.2.1 Environnement de travail
L’entraînement du model EfficientNetB1 a été réalisé sur un environnement sans GPU, ce qui pose certaines contraintes en termes de performance (en particulier sur la mémoire vive). Afin d’entraîner un modèle efficace malgré ces limitations, tout en optimisant l’usage des ressources disponibles. Une astuce fournit par Google est utilisé pour permettre un entrainement sur CPU, en configurant la fonction expérimental AutoShardPolicy.
3.2.2 Préparation à la prédiction
Les labels des images ont été récupérés à partir de .class_names, fournissant ainsi une liste ordonnée des classes.
Une fonction Python personnalisée get_champi_name() a été utilisée pour organiser les noms des labels en fonction des besoins spécifiques du projet pour la prédiction.
3.2.3 Entrainement du modèle
Les images d’entrée ont été redimensionnées en 224x224 pixels, conformément aux attentes du modèle pré-entraîné. Ce format est communément utilisé pour des modèles basés sur ImageNet.
Le modèle utilisé est pré-entraîné sur le dataset ImageNet, qui contient plus de 14 millions d’images et 1000 classes. Ce modèle, comprenant environ 9 millions de paramètres, permet d’obtenir une base solide pour la classification des images.
Le batch_size est fixé à 32, un compromis entre la vitesse d’entraînement et l’utilisation de la mémoire vive. La structure du modèle a été adaptée pour intégrer la couche finale de classification avec activation softmax, en fonction du nombre de classes cibles.
3.2.4 Évaluation des résultats
Sur 23 classes, le modèle a atteint une précision moyenne de 98 % lors de l’entraînement, indiquant une forte capacité à généraliser sur les données d’entraînement.
Les résultats sur les données de validation varient entre 80 % et 90 %, en fonction des ajustements apportés aux hyperparamètres. Cela montre que le modèle a une bonne capacité de généralisation, mais pourrait être affiné pour éviter le surapprentissage.
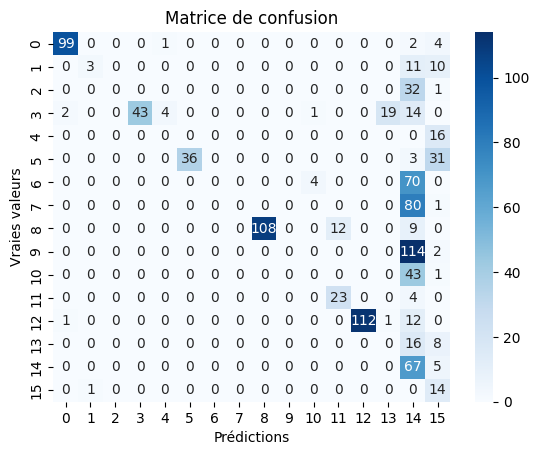
Certaines classes de champignion sont visiblement très problèmatique avec ce modèle et ne parviennent quasiment jamais à effectuer des prédictions juste.
Il est noté que certaines espèces de champignons, comme par exemple le Stropharia ambigua (classe 14) est souvent prédite comme une autre espece, la seule nuance qui permette de différencier étant la couleur jaunâtre propre a cette espèce, nous pouvons en déduire que ce modèle n’est pas très performant sur la prise en compte des nuances de couleurs.
Les Stropharia ambigua sont prédites sans prendre en compte leur couleur jaunâtre.
3.2.5 Optimisation
De nouveaux essais sont effectuées sur 16 classes uniquement pour volontairement exclure les classes problématiques, avec une augmentation des donnés et un nombre d’epoch plus généreux.
Ajout de callbacks : ReduceLROnPlateau pour améliorer la décente de gradient et EarlyStopping pour eviter le sur-entrainement.
Deux couches Dropout avec un taux de 0.5 ont est ajoutés au réseau pour le rendre plus robuste.
Les précédents résultats montrent que les predictions sont clairement moins fiables sur les dernières classes. Ceci est causé car les données ne sont pas mélangés aléatoirement sur les différents jeux de donné. Ainsi, un Shuffle est activé pour forcer l’entrainement des données dans un ordre aléatoire.
L’entrainement s’arrête après seulement 4 epochs grâce au EarlyStopping, le sur-entrainement sur ce modèle interevient très rapidement de par sa nature, mais offre de bonnes perfomances.
3.2.6 Conclusion
L’entraînement du modèle EfficientNetB1 sur un environnement sans GPU a permis d’obtenir des résultats satisfaisants malgré les limitations matérielles. En optimisant l’utilisation des ressources, notamment grâce à l’astuce de la configuration AutoShardPolicy, le modèle a pu tirer parti d’un environnement CPU tout en maintenant de bonnes performances.
L’utilisation d’un modèle pré-entraîné sur ImageNet fourni une base solide pour la classification. De plus, la gestion personnalisée des labels a permis une adaptation efficace aux besoins spécifiques du projet. Nous constatons cependant que ce modèle n’est malheureusement pas très performant lorsqu’il s’agit de nuancer les couleurs des différentes éspèces.
Les performances du modèle ont montré une précision d’entraînement remarquable à 96% et une précision de validation de 86%.
Sur le jeu de test, les scores sont cependant plus interessants :
| Accuracy | Precision | Recall | F1-score |
|---|---|---|---|
| 0.9286764705882 | 0.9336224871829 | 0.9286764705882 | 0.9290201971718 |
Bien que ces résultats soient encourageants, ils révèlent également des marges de progression, notamment pour affiner les scores de précision sur le jeu d'évaluation.
Ces conclusions ouvrent la voie à des pistes d’amélioration, telles que l’optimisation des hyperparamètres et une meilleure gestion des données pour minimiser le risque de sur-apprentissage, EfficientNetB1 étant particulièrement senssible au sur-entrainement.
Bien que l’entrainement sur CPU est satisfaisant, effectuer ces expérimentations avec un GPU devrais offrir un gain de vitesse.
3.3 ResNet50
Après avoir exploré EfficientNetB1, nous avons décidé de tester ResNet50. Cette architecture se distingue par ses blocs résiduels qui facilitent l’entraînement de réseaux très profonds en ajoutant des connexions directes entre les couches. Pour la reconnaissance des champignons, ResNet50 peut être particulièrement intéressant en raison de sa capacité à extraire des caractéristiques complexes tout en maintenant une efficacité computationnelle, ce qui est crucial pour des tâches de classification fines comme celle-ci.
3.3.1. Modèle de base
Le modèle est basé sur ResNet50, pré-entraîné sur le jeu de données ImageNet. Nous avons enlevé la partie supérieure du modèle (le “top”) pour adapter le réseau pré-entraîné à notre tâche spécifique de reconnaissance des champignons. La partie supérieure d’un modèle pré-entraîné est généralement conçue pour des classes spécifiques du jeu de données d’origine, comme ImageNet. En retirant cette partie, nous pouvons ajouter des couches adaptées à notre propre ensemble de classes, ce qui permet au modèle de s’ajuster aux spécificités de notre tâche de classification multiclasse. Nous avons ajouté une couche de GlobalAveragePooling2D suivie d’une couche Dense de 1024 neurones (taille couramment utilisée dans de nombreux réseaux de neurones pour les couches cachées) avec activation ReLU. La dernière couche de sortie est une couche Dense avec autant de neurones que de classes dans les données, utilisant une activation softmax pour la classification multiclasse.
Les couches du modèle pré-entraîné ResNet50 ont été gelées (non-entraînables) pour conserver les poids appris précédemment et éviter de modifier ces paramètres durant l’entraînement. Le modèle a été compilé avec l’optimiseur Adam et une faible valeur d’apprentissage (learning rate = 1e-4). La perte utilisée est categorical crossentropy, avec une métrique d’évaluation sur la précision.
Résultats obtenus :
Précision d’entraînement : Le modèle montre une précision qui commence à 71 % et atteint presque 100 % (99,96 %) à la fin de l’entraînement. Cela montre que le modèle apprend très bien les données d’entraînement, mais cela suggère aussi un risque de surapprentissage (overfitting).
Précision de validation : La précision de validation commence relativement élevée à 81 %, mais fluctue au fil des époques, se stabilisant autour de 84 %. Le modèle généralise relativement bien, mais ne montre pas d’amélioration significative après quelques itérations, suggérant un plateau dans l’apprentissage.
Perte de validation : La perte de validation diminue légèrement au début, mais à partir de la cinquième époque, elle commence à augmenter. Cela reflète encore une fois un surapprentissage, car la perte d’entraînement continue de baisser tandis que la perte de validation augmente. Cela signifie que le modèle se spécialise trop sur les données d’entraînement et ne parvient pas à bien généraliser sur de nouvelles données.
3.3.2. Modèles ajustés
-
Ajout de Dropout (0.5) Le Dropout a été ajouté après la couche de GlobalAveragePooling2D et après la couche Dense, avec un taux de 0,5. Cela permet de réduire le surapprentissage (overfitting) en désactivant aléatoirement 50 % des neurones pendant l’entraînement. Cela rend le modèle moins dépendant de certains neurones spécifiques et améliore sa capacité de généralisation.
Régularisation L2 (0.001) Une régularisation L2 a été appliquée sur la couche Dense. Cette technique pénalise les poids excessivement élevés, contribuant à réduire le surapprentissage en encourageant des poids plus petits. Cela aide à créer un modèle plus stable et capable de mieux généraliser aux nouvelles données.
Résultats : La précision d’entraînement atteint 77 %, tandis que la précision de validation passe de 70 % à 80 % avec une perte de validation en baisse constante, montrant que la régularisation par Dropout et L2 aide à mieux généraliser et à réduire le surapprentissage.
-
Unfreezed layers
Les 10 dernières couches du modèle de base ResNet50 ont été “défigées” pour être entraînables, ce qui permet à ces couches d’affiner leurs poids pendant l’entraînement. L’apprentissage est effectué avec un taux d’apprentissage plus bas (1e-5) pour éviter une mise à jour trop rapide des poids, et ce sur 10 époques.
Résultats : Le surapprentissage est probable, car l’exactitude en entraînement est très élevée, mais l’exactitude en validation stagne et la perte en validation ne diminue pas significativement.
-
Régularisation et Dropout : Deux couches de Dropout à 50% et la régularisation L2 sont ajoutées pour limiter le surapprentissage, en réduisant la dépendance du modèle à certaines connexions spécifiques.
Optimisation de l’entraînement Early Stopping : Le modèle arrête l’entraînement si la perte en validation ne s’améliore plus après 3 époques, tout en restaurant les meilleurs poids, évitant un surapprentissage inutile.
Réduction du taux d’apprentissage (ReduceLROnPlateau) : Ce callback diminue progressivement le taux d’apprentissage si la validation stagne, permettant des ajustements plus fins dans les dernières étapes d’entraînement.
Augmentation des epochs : Le nombre d'époques est passé à 50, en combinaison avec les mécanismes de contrôle du surapprentissage, pour permettre au modèle d’explorer un espace plus large de solutions tout en conservant une bonne généralisation.
Résultats : L’exactitude d’entraînement dépasse 94%, mais l’exactitude de validation progresse lentement et se stabilise, indiquant un possible surapprentissage. La perte d’entraînement diminue, tandis que la perte de validation augmente après quelques époques, renforçant l’hypothèse de surapprentissage. Le taux d’apprentissage, initialement à 1e-5, a été réduit à 2e-6 après la 7e époque, montrant que le modèle a atteint un plateau tôt.
-
Augmentation du taux de dropout à 0,7 pour réduire la dépendance excessive aux neurones spécifiques et prévenir le surapprentissage. 5 couches de ResNet50 sont désormais dégelées pour affiner davantage l’apprentissage. Le callback EarlyStopping a une patience augmentée à 5 pour permettre au modèle de continuer l’entraînement plus longtemps avant d’arrêter si la validation ne s’améliore pas, et le ReduceLROnPlateau réduit le taux d’apprentissage plus progressivement, avec un plancher fixé à 1e-6. Augmentation de la régularisation L2 dans la couche de sortie à 0.01 pour mieux contrôler le surapprentissage.
Résultats L’exactitude d’entraînement atteint 95%, mais la précision de validation stagne autour de 80%, suggérant un surapprentissage. La perte de validation diminue au début mais augmente ensuite, ce qui confirme également un surapprentissage. Le taux d’apprentissage réduit considérablement après la 12e époque, montrant que le modèle a atteint un plateau précoce dans l’entraînement.
Conclusion
Malgré une haute précision d’entraînement (95%), le modèle montre encore du surapprentissage, avec une précision de validation stagnante autour de 80% et une perte de validation croissante.
Causes possibles :
- Le modèle pré-entraîné sur ImageNet peut ne pas capturer suffisamment les caractéristiques spécifiques des champignons.
- Les données d’entraînement pourraient être insuffisantes pour une généralisation efficace.
- Le taux d’apprentissage pourrait ne pas être optimal pour ce problème spécifique.
Prochaines étapes :
- Augmenter la taille et la diversité des données d’entraînement sur les champignons.
- Ajuster ou simplifier l’architecture du modèle.
- Ajouter des techniques de régularisation supplémentaires.
- Expérimenter avec des modèles spécifiquement entraînés sur des données de champignons ou utiliser des techniques de transfert learning adaptées.
3.4. ResNet18
ResNet18 est comme ResNet50 un réseau de neurones convolutifs utilisant des connexions résiduelles, mais avec une profondeur de 18 couches seulement.
Les essais menés avec ResNet18 ont été immédiatement concluants avec un score de précision d’environs 97% sur notre dataset (23 classe avec en moyenne 166 images par classe).
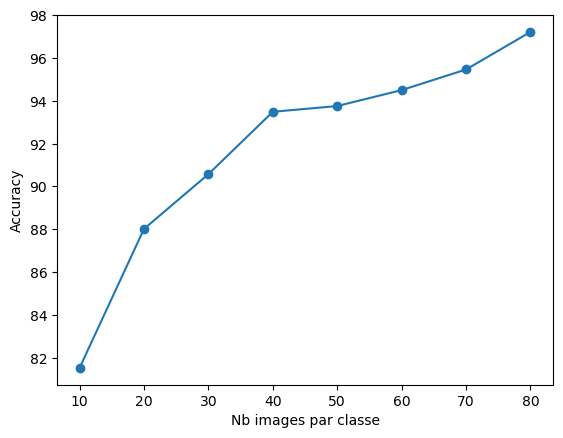
Différents essais on permis d’estimer le nombre d’images necessaires pour obtenir un niveau de précision satsfaisant. Ainsi on constate qu’avec un dataset comprenant seulement 80 images par classe on atteint une précision de 97%. Avec seulement 30 images par classe on reste au dessus de 90% de précision, et même avec seulement 10 images par classe on reste au dessus de 80% de précision.
3.5 JarviSpore
Suite aux résultats mitigés sur le transfert Learning, nous avons pris l’initiative de créer un modèle de zéro avec comme support les cours DataScientest et des livres.
Nous avons investi dans un PC avec carte graphique RTX 3090 disposant de 24 Go de VRAM. Notre PC dispose de 192 Go de RAM. Processeur i9 14900k.
Nous avons ensuite pris le parti de tester dans un environnement WSL2. Cela nous permettait d’utiliser les dernières versions de TensorFlow, Cuda, Cudnn et Keras.
Après l’installation, nous avons construit le modèle dans VSCode, mais lors des entraînements, des problèmes de mémoire nous ont compliqués la tâche.
Nous avons déployé un environnement sous Windows en utilisant d’anciennes versions de TensorFlow, Cuda …
Pour assurer la compatibilité des bibliothèques utilisées et de leurs versions, car la compatibilité TensorFlow sous Windows s’arrête à la version 2.10 :
numpy : 1.26.4
tensorflow : 2.10.0
matplotlib : 3.9.2
scikit-learn : 1.5.2
PIL : 10.4.0
cv2 : 4.10.0
pandas : 2.2.3
Ce modèle effectue l’entraînement, l'évaluation et l’interprétation d’un modèle de réseau de neurones convolutif (CNN) pour une tâche de classification d’images. Voici les différentes étapes et le processus utilisés :
-
Importation des Bibliothèques
Nous commençons par importer les bibliothèques nécessaires pour la manipulation des données, l’entraînement du modèle, l'évaluation et la visualisation des résultats. Les bibliothèques incluent TensorFlow pour la construction du modèle, NumPy pour les calculs numériques, Pandas pour la gestion des données et OpenCV pour le traitement des images. -
Extraction des Versions des Bibliothèques
Nous vérifions les versions des bibliothèques utilisées afin d’assurer la compatibilité des versions. -
Chargement des Datasets (structurées et non structurées)
Nous définissons les chemins pour les datasets d’entraînement, de validation et de test. Nous utilisons la fonction image_dataset_from_directory pour charger les images en les redimensionnant à la taille (224, 224) avec un batch size de 32 images. Les ensembles de données sont ensuite configurés pour être mis en cache en mémoire vive, préchargés et optimisés. -
Chargement des Classes
Nous chargeons les noms des classes à partir d’un fichier CSV (API MushroomObserver) pour obtenir la liste des classes disponibles. Cela permet au modèle d’associer les indices des classes avec les noms réels lors de l’affichage des résultats. -
Construction du Modèle Convolutionnel
Nous construisons un CNN personnalisé avec plusieurs couches de convolution suivies de la normalisation par lots (Batch Normalization), du sous-échantillonnage (MaxPooling) et d’une couche de sortie utilisant softmax pour la classification des 23 classes. Les couches de convolution permettent d’extraire les caractéristiques des images, tandis que les couches denses à la fin effectuent la classification. -
Compilation du Modèle
Le modèle est compilé avec l’optimiseur Adam et la fonction de perte sparse_categorical_crossentropy, adaptée à la classification multi-classes avec des étiquettes sous forme d’entiers. -
Ajout de l’Early Stopping et du Model Checkpoint
Nous configurons des callbacks pour arrêter l’entraînement si la précision de validation n’augmente plus après 5 époques (early stopping) et pour sauvegarder le meilleur modèle lors de l’entraînement (ModelCheckpoint). -
Gestion du Déséquilibre des Classes
Nous vérifions le déséquilibre des classes dans l’ensemble d’entraînement. Si certaines classes sont moins représentées, nous utilisons des pondérations de classe (class_weight) pour accorder plus d’importance aux classes sous-représentées afin d’améliorer la généralisation du modèle. -
Entraînement du Modèle
Le modèle est entraîné sur 20 époques, en utilisant les pondérations de classe pour mieux gérer les déséquilibres. Les callbacks configurés permettent de surveiller la performance et de sauvegarder le meilleur modèle. -
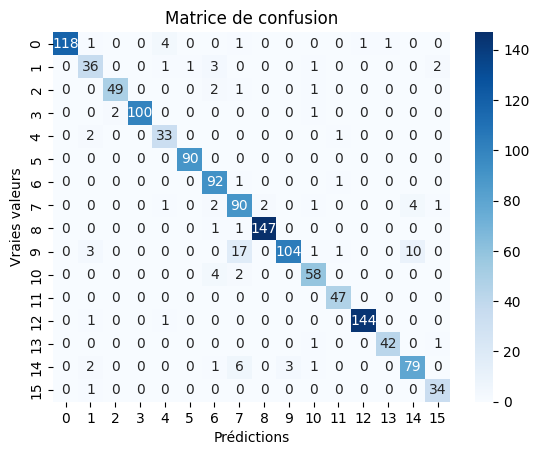
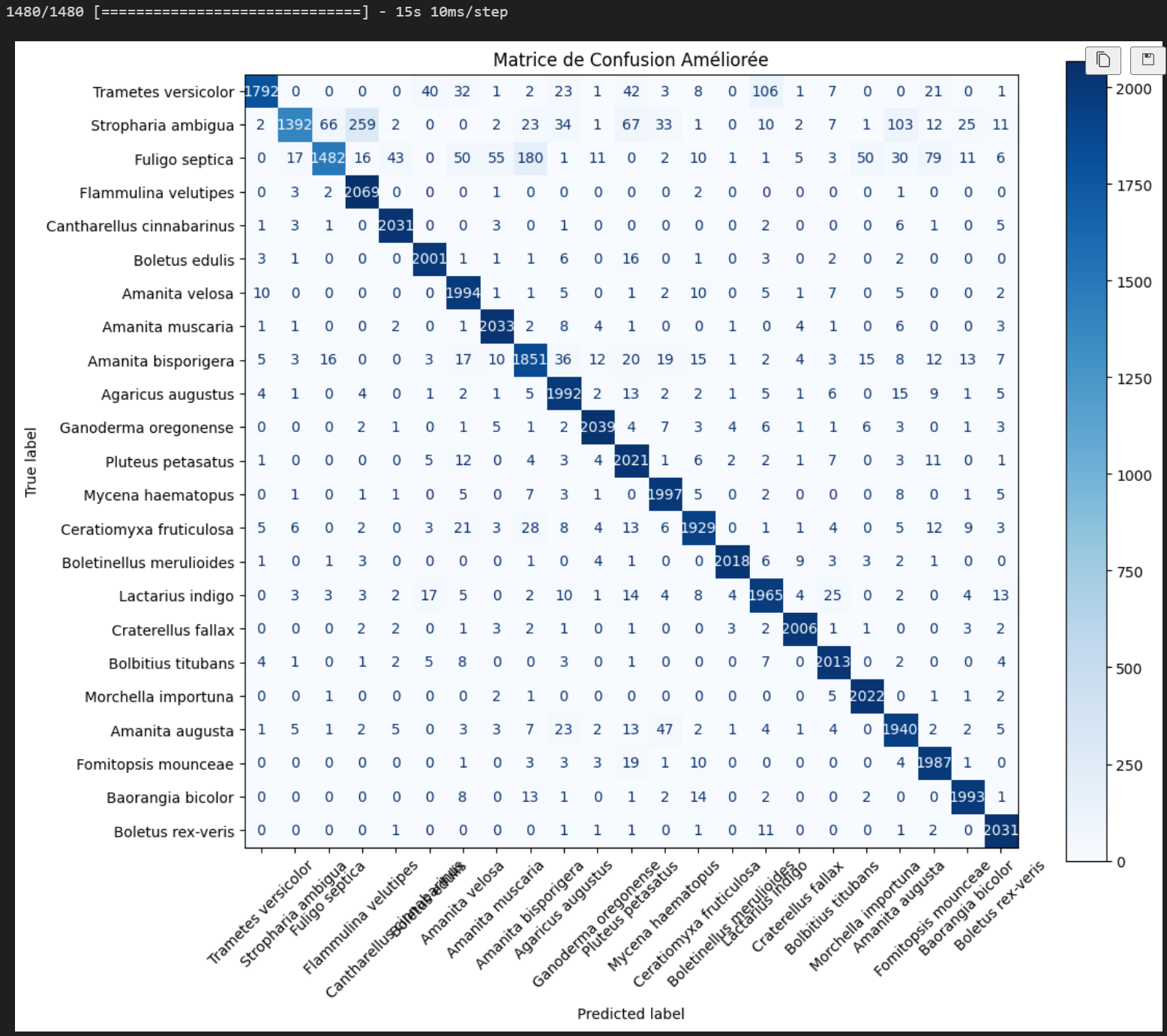
Génération de la Matrice de Confusion
Après l’entraînement, nous générons une matrice de confusion sur l’ensemble de validation pour évaluer la capacité du modèle à classifier correctement les images. La matrice de confusion est affichée avec les noms des classes pour faciliter l’interprétation des résultats. -
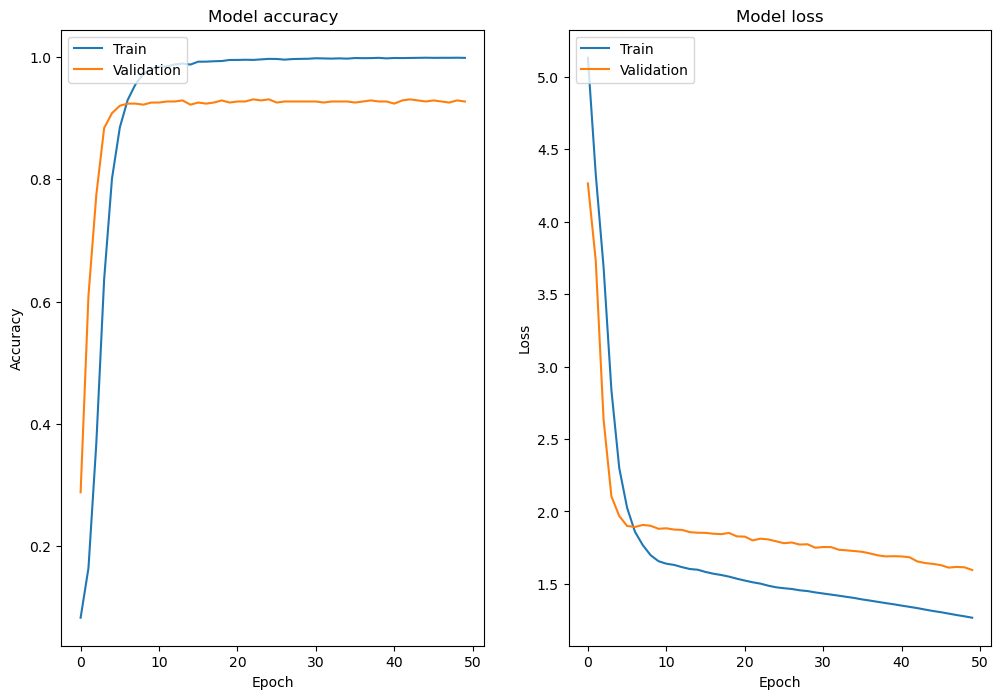
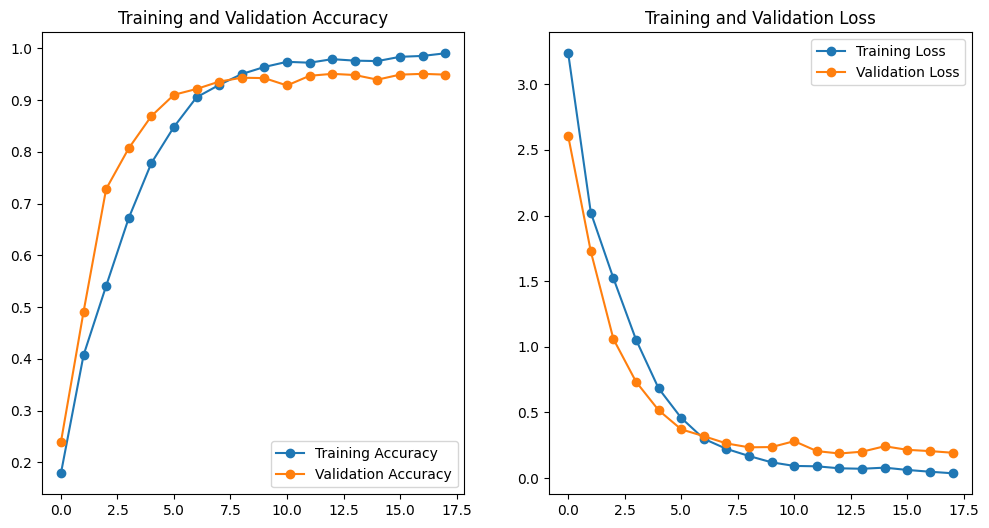
Visualisation des Courbes d’Entraînement
Nous affichons les courbes de précision et de perte pour les ensembles d’entraînement et de validation, ce qui nous permet de visualiser l'évolution des performances du modèle pendant l’entraînement. -
Sauvegarde du Modèle et Métadonnées
Nous sauvegardons le modèle entraîné au format .keras ainsi que les métadonnées (date d’entraînement, précision sur l’ensemble de test, nombre d'époques). Cela permet de documenter le modèle pour un suivi ultérieur. -
Test et Évaluation du Modèle sur l’Ensemble de Test
Nous testons le modèle sur le jeu de données de test pour obtenir la précision finale et évaluer sa performance générale. -
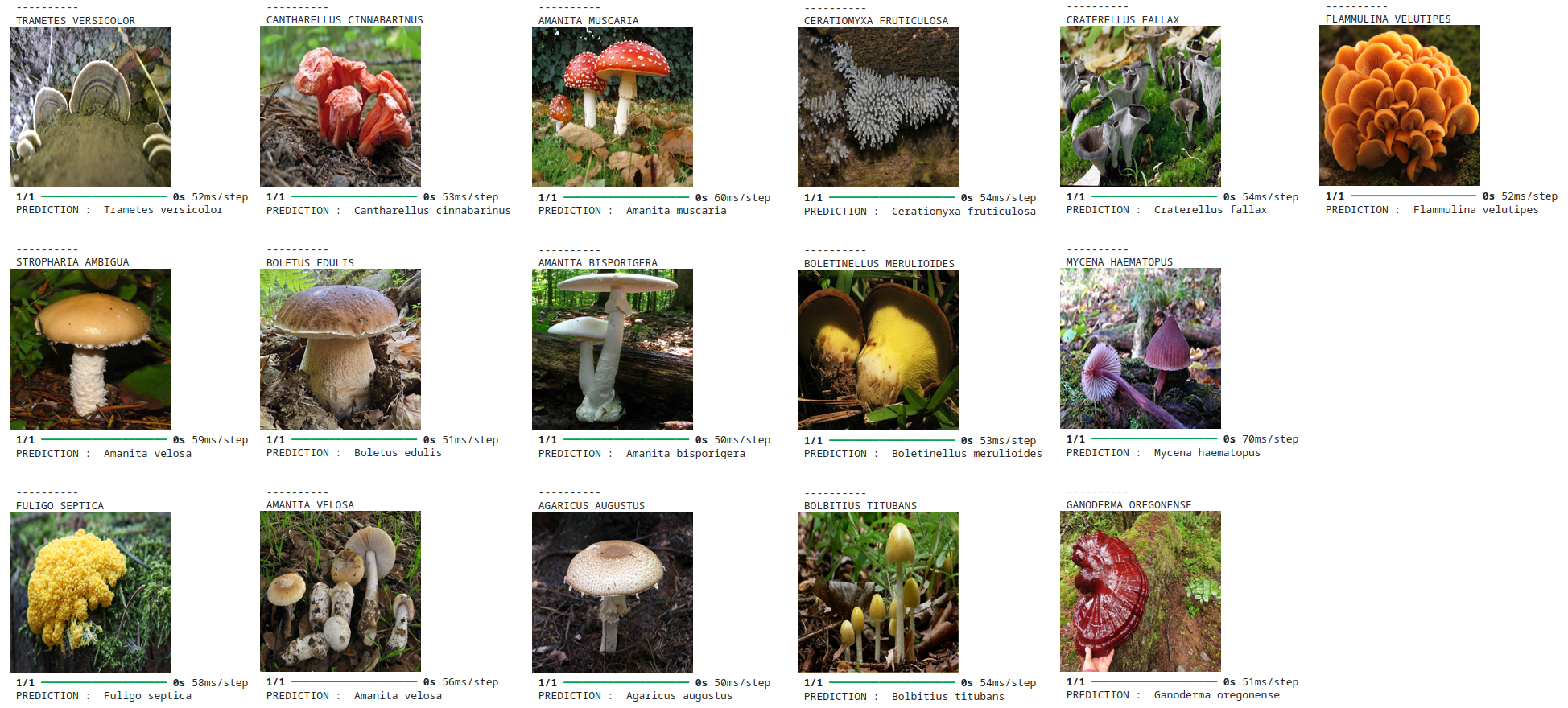
Affichage Grad-CAM
Nous implémentons Grad-CAM pour visualiser les activations des couches de convolution du modèle. Cette technique permet d’afficher les régions de l’image qui ont le plus contribué à la décision du modèle. Les résultats sont affichés pour cinq images aléatoires du jeu de test.
Résultats Attendues
- Précision du Modèle : La métrique mesurée est la précision, elle permet de mesurer le pourcentage de classifications correctes effectuées.
- Interprétabilité avec Grad-CAM : Les heatmaps générées par Grad-CAM doivent indiquer les parties pertinentes de l’image, ce qui aide à comprendre le fonctionnement du modèle.
- Généralisation : Avec l’utilisation des callbacks et des pondérations de classe, le modèle doit éviter le sur-apprentissage et bien généraliser sur les données de validation et de test.
Ces étapes permettent de construire un modèle performant pour la classification d’images, tout en prenant en compte les déséquilibres de classe et en offrant des outils d’interprétation des résultats.
Lien vers le modèle sur Hugging Face : https://huggingface.co/YvanRLD/JarviSpore
4. Interprétation des résultats avec Grad-CAM
Pour mieux comprendre les résultats et les décisions prises par les algorithmes, nous avons utilisé Grad-CAM (Gradient-weighted Class Activation Mapping), une technique puissante d’interprétation des modèles de deep learning, en particulier pour la classification d’images. Cette méthode permet de visualiser les régions d’une image qui influencent le plus les décisions d’un modèle. En générant des cartes thermiques (heatmaps) superposées sur les images d’entrée, Grad-CAM met en évidence les caractéristiques jugées essentielles par le modèle pour ses prédictions.
Pour créer ces cartes thermiques, on commence par calculer les gradients associés à la classe prédite, en les reliant aux cartes de caractéristiques issues de la dernière couche de convolution. Ces gradients sont ensuite moyennés pour obtenir une vue d’ensemble, qui sert à ajuster les cartes de caractéristiques, mettant ainsi en lumière les zones les plus importantes pour la classification.
Avec Grad-CAM, nous pouvons mieux comprendre les performances de nos modèles en analysant visuellement leurs points d’attention. Cette approche nous aide à identifier les forces et les faiblesses des modèles, à déceler d'éventuels biais et à approfondir notre compréhension des décisions prises par les algorithmes.
Le graphique ci-dessous illustre des exemples de cartes thermiques obtenues via EfficientNetB1 et ResNet50 pour quatre classes différentes de champignons.
Les “zones chaudes” (zones rouges et jaunes des cartes thermiques) indiquent les régions sur lesquelles le modèle se concentre. En général, ces zones chaudes correspondent à certaines parties du champignon, mais la zone de concentration varie selon la classe de champignon (par exemple, la tige par rapport à la tête du champignon, le bord de la tête, etc.). Il est intéressant de noter que, pour l’image contenant deux champignons, ResNet50 performe mieux en identifiant les deux, tandis qu’EfficientNet se concentre principalement sur un seul champignon. Cependant, pour la photo avec la présence de la main, ResNet50 était complètement perdu et ne se concentrait pas du tout sur le champignon, tandis qu’EfficientNet l’identifiait mieux.
En somme, ces résultats soulignent l’importance d’une analyse approfondie pour mieux comprendre les performances de chaque modèle dans des situations variées.
5. Conclusion
5.1 Comparaison des resultats
Les différents essais réalisés ont mis en évidence d’importantes différences de résultats obtenus avec divers modèles sur un même jeu de données. Alors que certains modèles, comme VGG16, affichent des limites significatives pour ce cas d’utilisation, d’autres, tels que ResNet18, ont démontré d’excellentes performances.
En poursuivant notre analyse, nous avons également comparé ResNet18 et ResNet50. Cette comparaison montre qu’un modèle plus profond n’est pas toujours synonyme de meilleures performances ; au contraire, sur un petit jeu de données, un modèle plus complexe peut s’avérer moins performant.
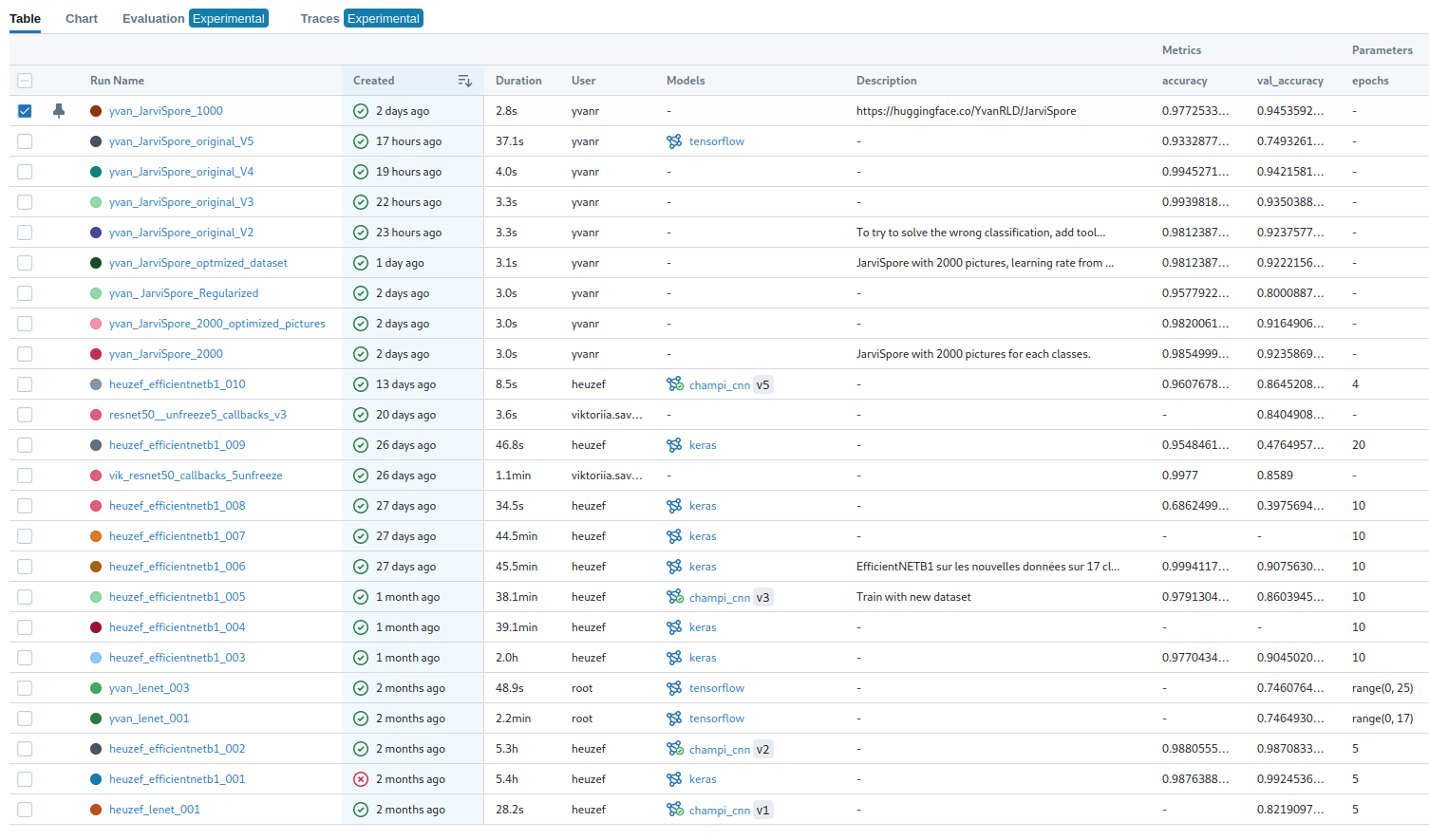
Dans le cadre de notre projet, nous avons intégré l’approche MLflow pour améliorer le suivi et la gestion de nos expériences en apprentissage automatique.
Ce dernier est utilisé pour tracer chaque étape du processus expérimental, notamment les paramètres, les métriques, et les artefacts des modèles.
Nous avons configuré un serveur de tracking, défini un projet spécifique pour organiser nos expérimentions.
Cette intégration permet de centraliser et comparer les métriques et de faciliter le déploiement ultérieur des modèles retenus.
Ainsi, nous pouvons suivre de manière systématique et efficace les progrès réalisés dans notre projet. Les captures suivantes résument donc les résultats de l’ensemble du projet :
5.2 Interpretabilité
Les différentes évaluation des modèles effectués sur des données de tests montrent que de façon global, les modèles ont tous tendance à effectuer de la sur-interprétation et gèrent particulièrement mal les couleurs des champignons.
En effet les méthodes Grad-Cam permettent de visualiser cette tendance à prendre en compte des zones précises, sans se concentrer sur les zones très colorés. La couleur est pourtant l’un des points les plus importants, les modèles montrent tous de grandes faiblesse pour différencier deux champignons physiquement identique avec la couleur comme seul élément de différenciation ou encore de simplement localiser un champigon, même de couleur vive, si le fond de la photo contient des éléments avec une forte luminosité proche du blanc.
5.3 Technique
Nous pouvons noter que si les modèles avec une architeture les plus basiques (Lenet) offre des resultats très moyen, ceux en transfert learning offrent cependant des resultats performants même si rapidement sujet à un sur-apprentissage malgrès nos essais avec différentes technique d’optimisation.
Nous concluons sur le fait que la conception d’un modèle sur-mesure, avec une architecture complexe, bien que très fastidieux, permet d’obtenir des métriques et rapports de classification plus performant à tout les niveaux.
En effet, décision est prise d’implémenter un modèle nommé JarviSpore, solution modulable au fur et à mesure de l’avancée de nos connaissances. Celui-ci est arrivé à maturité et présente des performances supérieures.
6. Pour aller plus loin
Modèles Open-Source
L’utilisation de modèles plus récents accessible sur HuggingFace nous permettrais très surement d’obtenir encore de meilleurs performance de précision.
Nous expérimentons l’utilisation d’un modèle communautaire de classification pré-entrainé sur 100 espèces de champignons russe.
Ce modèle, partagé par Dmytro Iakubovskyi, est entrainé sur 233480 images et se base sur l’architecture ViT (85.9M de paramètres).
🤗 Mid-ViT par dima806 - HuggingFace
Réseaux Kolmogorov-Arnold
Les MLP (réseaux de neurones multicouches), bien qu’utilisés dans de nombreux contextes, sont souvent sujets à l’overfitting en raison de leur grande flexibilité, et comportent de nombreux paramètres difficiles à interpréter, ce qui limite leur utilité dans certaines applications.
Récemment, les réseaux Kolmogorov-Arnold (KAN) ont été proposés comme une alternative prometteuse (article : https://arxiv.org/abs/2404.19756, GitHub : https://github.com/KindXiaoming/pykan).
Contrairement aux MLP, qui utilisent des fonctions non linéaires fixes comme ReLU ou Tanh, les KAN exploitent des B-splines, des polynômes par morceaux, pour modéliser les données de manière plus souple et ajustée. Cela permet d’améliorer l’interprétabilité des modèles et de réduire le nombre de paramètres, rendant les KAN plus efficaces et potentiellement moins sensibles à l’overfitting.
Cependant, bien que les KAN présentent de nombreux avantages théoriques, ils restent instables, avec des résultats sensibles aux hyperparamètres choisis, ce qui nécessite des ajustements soigneux pour chaque tâche.
Pour les prochains tests, il sera crucial d’explorer davantage cette nouvelle architecture, de tester son potentiel de généralisation sur des données variées, et d'évaluer dans quelle mesure elle peut remplacer les MLP dans des architectures complexes.